对于已经拟合好的生存模型,我们一般会直接用ROC去评判一下整体的水平,因为很多时候阈值都是我们人为根据实际情况去设定的,这种微调的细节都是在整体模型的拟合程度确定下来后再做的工作。
ROC曲线可以提供给我们模型对于二分类变量的区分能力,而且还可以看到到底是哪些样本数据被错误分类了,能帮助我们确定哪些数据可能有共线性或迷惑性的特征。
以下是一个例子:
# 加载必要的包
library(pROC)
library(ggplot2)# 生成模拟数据集
set.seed(123)
n <- 1000# 生成两个正态分布的组
group1 <- rnorm(n, mean = 0, sd = 1) # 负例
group2 <- rnorm(n, mean = 1.5, sd = 1) # 正例# 创建数据框
data <- data.frame(score = c(group1, group2),class = factor(rep(c(0, 1), each = n))
)# 计算ROC曲线
roc_obj <- roc(data$class, data$score)# 获取AUC值
auc_value <- auc(roc_obj)
cat("AUC值为:", auc_value, "\n")# 找到最佳阈值(根据Youden指数)
best_threshold <- coords(roc_obj, "best", ret = "threshold", best.method = "youden")# 修正后的阈值输出方式(提取数值部分)
cat("最佳阈值为:", best_threshold$threshold, "\n")# 或者使用更安全的方式
if(!is.null(best_threshold$threshold)) {cat("最佳阈值为:", best_threshold$threshold, "\n")
} else {cat("无法确定最佳阈值\n")
}# 计算所有阈值下的指标
roc_table <- coords(roc_obj, "all", ret = c("threshold", "specificity", "sensitivity", "accuracy"))
head(roc_table)# 绘制ROC曲线
ggroc(roc_obj, color = "blue", size = 1) +geom_abline(intercept = 1, slope = 1, linetype = "dashed", color = "red") +labs(title = paste0("ROC曲线 (AUC = ", round(auc_value, 3), ")"),x = "假正例率 (FPR)", y = "真正例率 (TPR)") +theme_minimal()输出:
AUC值为: 0.858621
最佳阈值为: 0.8868806 0.9051289threshold specificity sensitivity accuracy
1 -Inf 0.000 1 0.5000
2 -2.735349 0.001 1 0.5005
3 -2.652036 0.002 1 0.5010
4 -2.622424 0.003 1 0.5015
5 -2.554809 0.004 1 0.5020
6 -2.486908 0.005 1 0.5025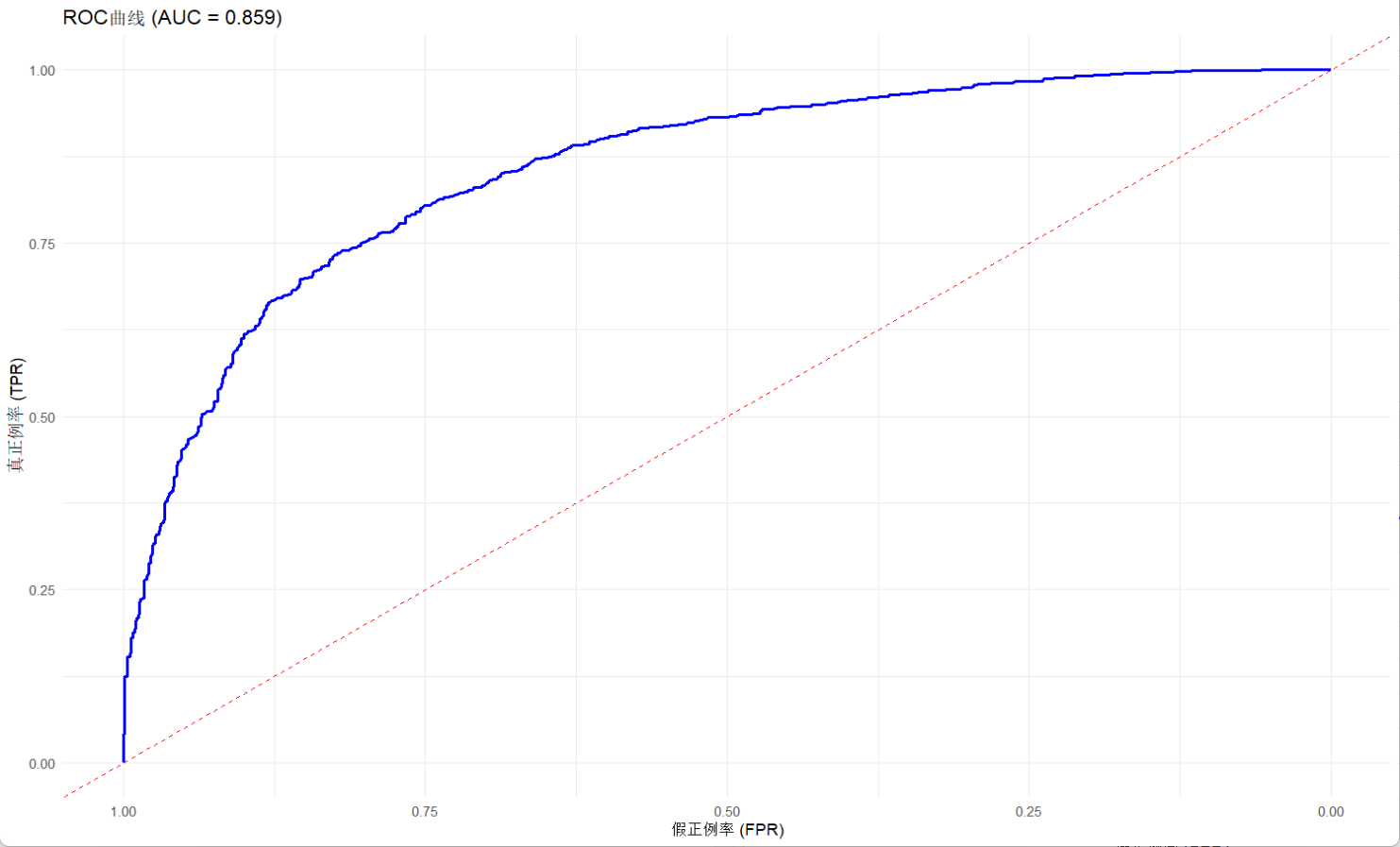
可以看到,曲线整体偏左上角,符合AUC靠近1的事实,说明模型对于二分类的区分能力较好,给出的最佳阈值虽然不一定准确,但是也接近这个范围,进一步说明其解释性。




:封装自定义可复用组件)











:从原理到现代实现演进)


