一、概念
首先还是老规矩,处理一下概念问题,好将之后的问题进行分类和区分
单细胞转录组:指在单个细胞水平上对转录组(即细胞内所有转录出来的 RNA,主要是 mRNA)进行研究的学科或技术方向,核心是分析单个细胞中基因的表达情况(包括哪些基因被激活表达、表达量高低等),从而揭示细胞间的异质性、功能差异及分子调控机制。
可以简单理解为:研究 RNA——>揭示细胞内群体异质性、定义细胞类型、细胞状态和细胞的动态转变
空间转录组(ST):是一种结合空间位置信息和基因表达数据的技术,能够在组织切片上解析基因表达的空间分布模式,弥补了传统单细胞转录组技术丢失空间信息的缺陷。
空间转录组能够将基因表达信息与细胞在组织中的具体位置相结合,从而揭示细胞类型的分布、细胞亚群的空间组织以及细胞间的相互作用。
二、具体应用
这个部分只专注于实验设计和样本,不关注研究内容
案例一:scRNA-seq+ST破译结直肠癌干细胞异质性与转移路径
该研究通过scRNA-seq和空间转录组(ST)分析,揭示了结直肠癌中异质性癌症干细胞样细胞(CSCs)及其器官特异性转移的机制。

样本:8例原发性CRC、5例oCRC(卵巢转移)、2例lCRC(肝脏转移)、7例邻近正常组织和7例外周血单核细胞(PBMC),共29例样本。
研究路线: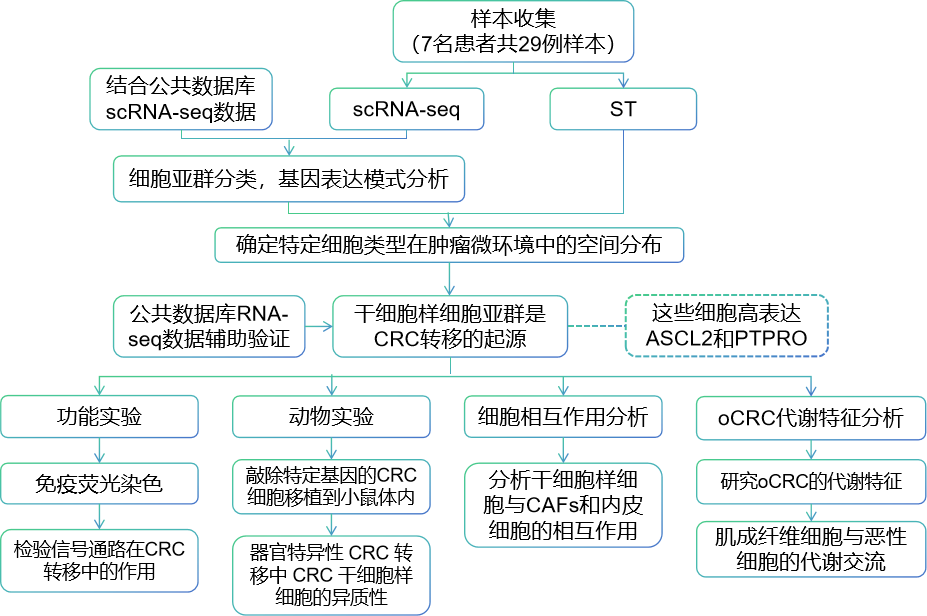
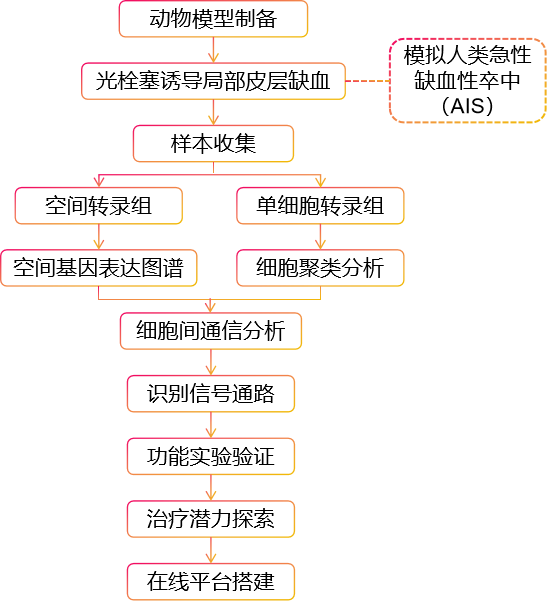
案例二:scRNA-seq+ST表征缺血小鼠大脑的分子和细胞结构
本研究利用空间转录组学和scRNA-seq技术对缺血性脑卒中小鼠模型的大脑进行了深入分析,揭示了脑卒中后神经炎症反应中的关键分子和细胞变化。

样本:接受光栓塞手术组和假手术对照组的两组小鼠的梗死周边区域
研究路线:
案例三:scRNA-seq+ST揭示HNSCC中CAF亚群限制了CD8+T细胞的浸润和抗肿瘤活性
本文为关于头颈部鳞状细胞癌(HNSCC)中癌症相关成纤维细胞(CAF)亚群对CD8+ T细胞浸润和抗肿瘤活性的影响。研究通过空间转录组和scRNA-seq技术,揭示了特定的CAF亚群与CD8+ T细胞浸润限制和功能障碍相关。这些CAFs表达高水平的CXCL9、CXCL10、CXCL12和MHC-I分子,并且富含半乳糖结合蛋白-9(Gal9)。
样本:来自HNSCC患者5个肿瘤组织和5个配对的邻近正常组织
研究路线:
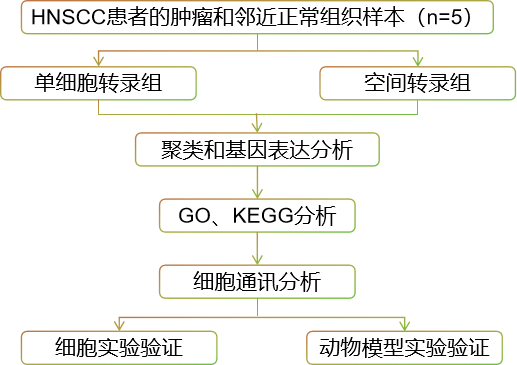
三、整合方法分析方法总结
在《Nature reviews genetics》发表综述文章,回顾了整合scRNA-seq与空间转录组学技术研究的尝试和努力,包括新兴的整合计算方法,并提出了有效结合当前方法的途径。
参考文献
Longo, S.K., Guo, M.G., Ji, A.L. et al. Integrating single-cell and spatial transcriptomics to elucidate intercellular tissue dynamics. Nat Rev Genet (2021). https://doi.org/10.1038/s41576-021-00370-8
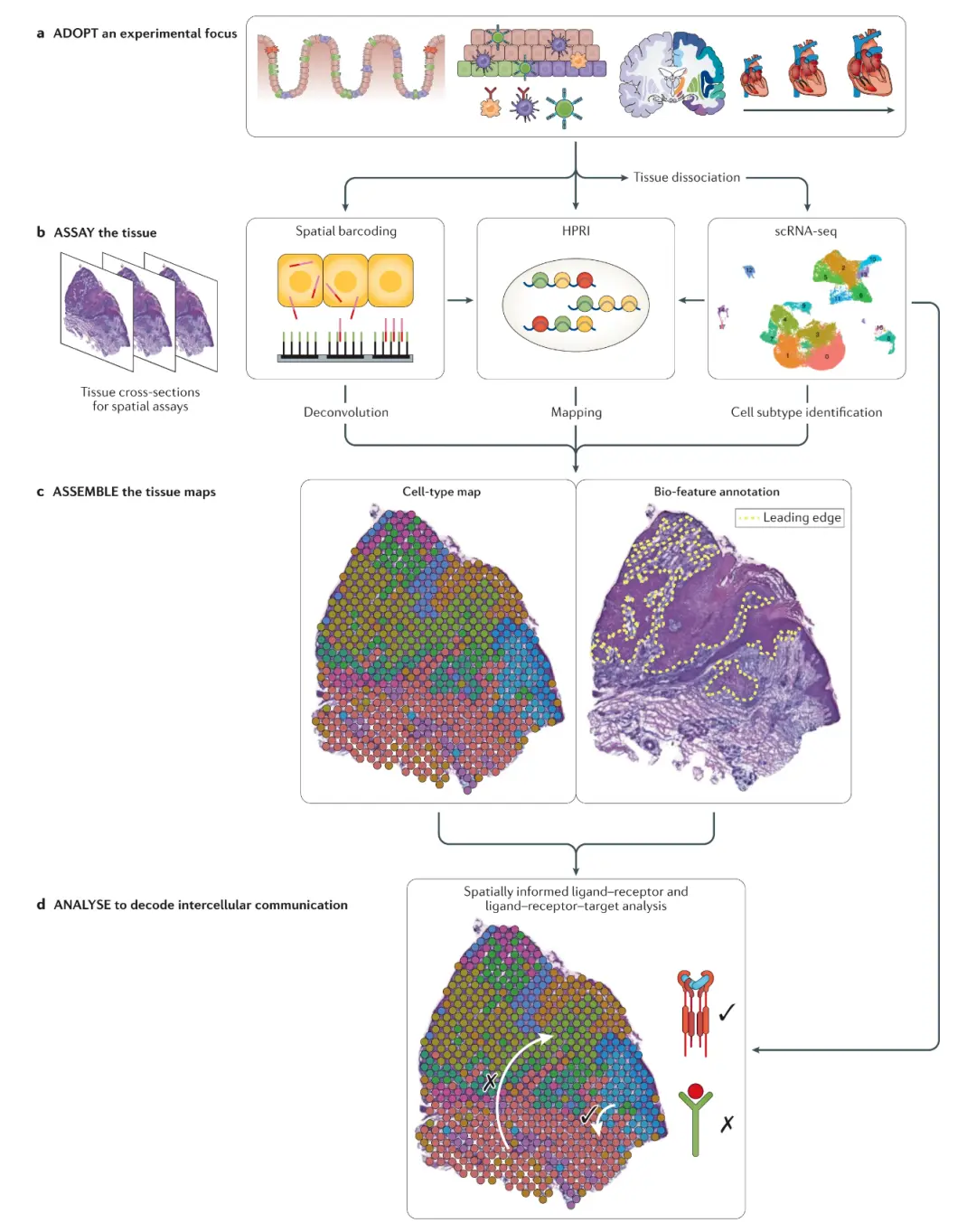
整合scRNA-seq和空间转录组学研究的流程模式
核心流程(a→b→c→d)
- a ADOPT:确定实验对象(如组织、器官、疾病模型),明确研究聚焦方向(分子、细胞、器官层面)。
- b ASSAY:组织分析环节,包含 3 类技术:
- Spatial barcoding:空间条形码技术(如 10X Visium),通过 “空间反卷积(Deconvolution)” 解析混合细胞的空间分布;
- HPRI:高分辨率原位分析(High - Plex RNA Imaging),实现基因表达的空间 “映射(Mapping)”;
- scRNA - seq:单细胞转录组测序,用于 “细胞亚型鉴定(Cell subtype identification)”。
- c ASSEMBLE:整合分析结果,生成 2 类图谱:
- Cell - type map:细胞类型空间分布图(标注不同细胞类型的区域);
- Bio - feature annotation:生物特征注释图(如标记 “Leading edge” 等功能区域)。
- d ANALYSE:基于空间图谱,解析 “细胞间通讯(intercellular communication)”,验证配体 - 受体互作对(Ligand - receptor pairs)及下游调控关系。
scRNA-seq+空间组学整合分析的研究进展(2021年止)
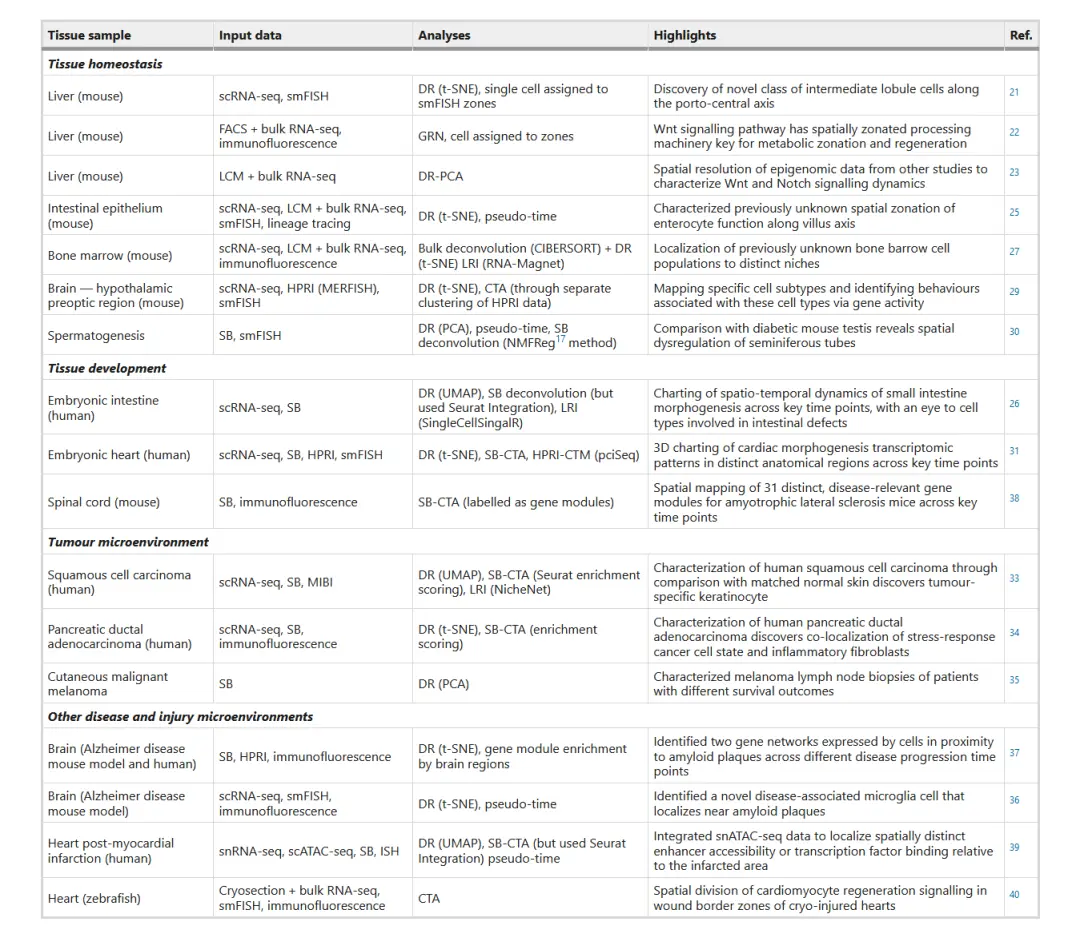
解析scRNA-seq和空间转录组数据的研究
核心维度(列)
- Tissue sample:研究对象(组织 / 疾病模型),分 5 大类:组织稳态(Tissue homeostasis)、组织发育(Tissue development)、肿瘤微环境(Tumour microenvironment)、其他疾病微环境(Other disease and injury microenvironments)。
- Input data:实验输入数据类型,如单细胞测序(scRNA-seq)、单分子荧光原位杂交(smFISH)、流式分选(FACS)、激光捕获显微切割(LCM)等。
- Analyses:分析方法,涉及降维(DR,如 t-SNE、UMAP )、基因调控网络(GRN)、反卷积(Deconvolution)、拟时序分析(pseudo-time )等。
- Highlights:研究亮点(核心发现),如揭示基因表达的空间分区(spatial zonation )、细胞亚型映射(cell subtypes mapping )、疾病相关分子网络(gene networks )等。
- Ref.:文献引用编号(对应研究的参考文献)。
研究场景覆盖
涵盖正常组织稳态(如肝脏、肠道)、发育(胚胎肠道、心脏)、肿瘤(鳞状细胞癌、胰腺癌)、疾病模型(阿尔茨海默病、心肌梗死) 等方向,体现空间组学在解析组织异质性、细胞功能、疾病机制中的应用。
scRNA-seq+空间组学整合分析的计算方法
整合scRNA-seq和空间转录组数据有两种主要方法:去卷积(Deconvolution)和映射(Mapping)。去卷积旨在根据单细胞数据,从每个捕获点的mRNA转录物的混合物中分离出离散的细胞亚群;映射有两方面:将指定的基于scRNA的细胞亚型定位到HPRI图谱上的每个细胞和将每个scRNA-seq细胞定位到组织的特定生态位或区域。
- 去卷积:被用于从混合细胞样本中分离出不同细胞类型的表达模式。
- HPRI(High-plex RNA Imaging) :高通量RNA成像技术,提供单细胞分辨率的空间数据,用于将scRNA-seq识别的细胞类型映射到组织中的实际位置。
- 映射:以单细胞分辨率创建空间分辨率的细胞类型映射。
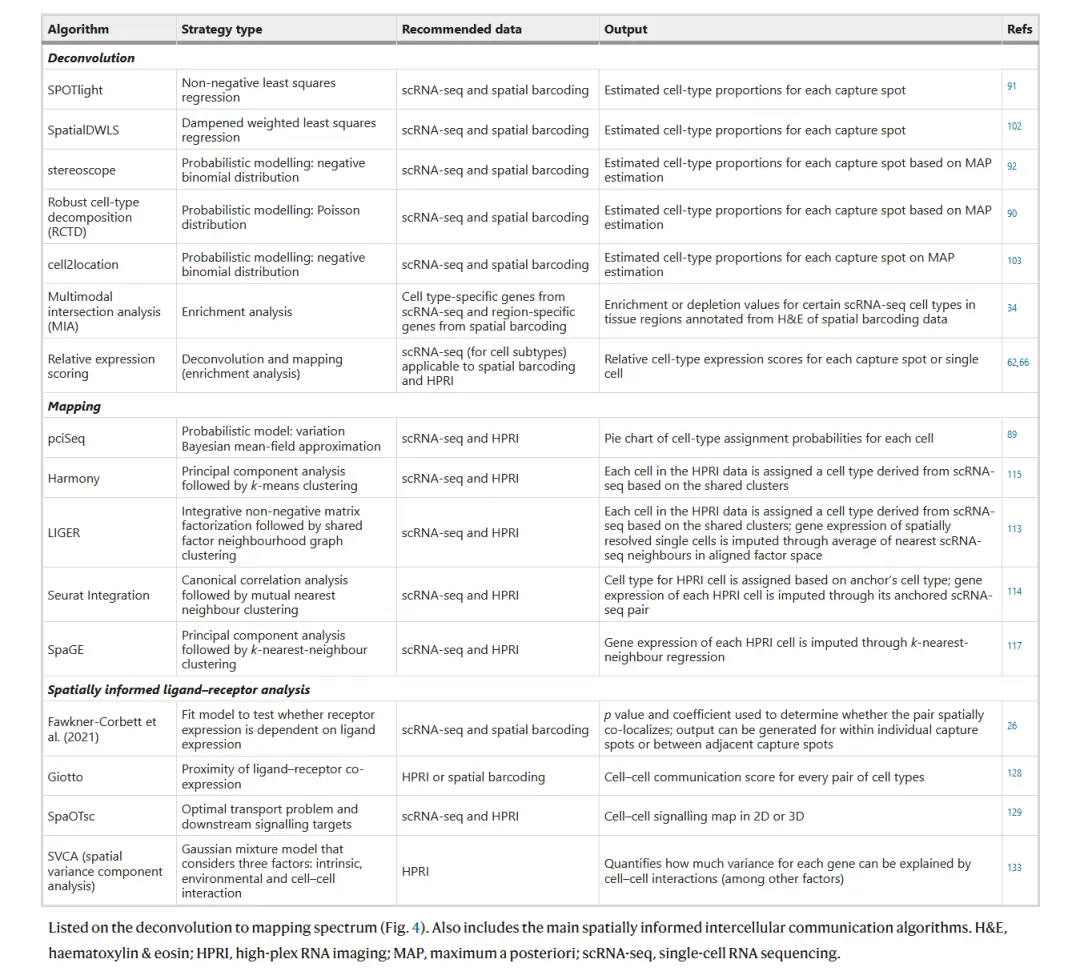
空间转录组与单细胞数据分析工具的分类对比表
核心维度(列)
- Algorithm:算法名称(如 SPOTlight、stereoscope 等),覆盖 3 类分析方向:
- Deconvolution(去卷积):解析混合信号的细胞类型比例(如 SPOTlight、SpatialDWLS );
- Mapping(映射):关联单细胞与空间数据(如 scSeq、Harmony );
- Spatially informed ligand-receptor analysis(空间配体 - 受体分析):解析细胞间通讯(如 SpaOTsc、Giotto )。
- Strategy type:算法策略(如 Non-negative least squares regression、Probabilistic modelling )。
- Recommended data:推荐输入数据类型(scRNA-seq、空间条形码数据、HPRI 等)。
- Output:工具输出结果(如细胞类型比例、配体 - 受体互作对 )。
- Refs:文献引用(对应工具的方法学或应用文献)。
价值与应用
这张表汇总了空间多组学分析的主流算法工具,覆盖去卷积、数据整合、细胞通讯 3 大核心场景,可用于指导空间转录组 + 单细胞数据分析的工具选型(如解析细胞类型比例选 SPOTlight,做细胞通讯选 SpaOTsc )。
单细胞和空间转录组数据的整合策略
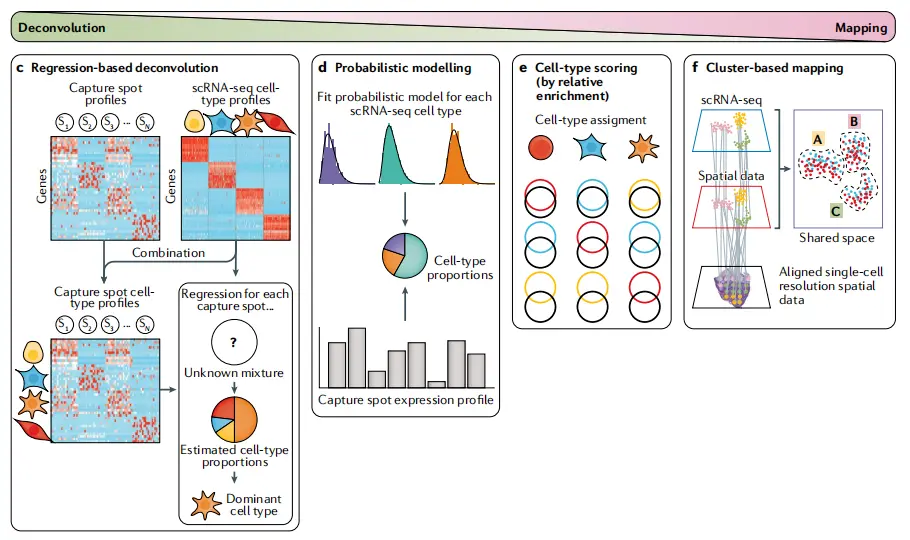
“去卷积(Deconvolution)与映射(Mapping)” 流程的示意图
模块拆解(左→右)
-
c Regression-based deconvolution(基于回归的去卷积):
用单细胞转录组的细胞类型表达谱(scRNA-seq cell-type profiles),通过回归模型(Regression)解析空间捕获点(Capture spot)的混合表达信号,估算细胞类型比例,识别 “主导细胞类型(Dominant cell type)”。 -
d Probabilistic modelling(概率建模去卷积):
为每种单细胞类型拟合概率模型(如分布曲线),结合空间捕获点的表达谱(Capture spot expression profile),推断细胞类型比例(Cell-type proportions)。 -
e Cell-type scoring(细胞类型评分映射):
通过 “相对富集(relative enrichment)” 给细胞类型打分,完成 “细胞类型分配(Cell-type assignment)”,用不同符号 / 颜色标记空间点的细胞类型归属。 -
f Cluster-based mapping(基于聚类的映射):
将单细胞转录组(scRNA-seq)与空间数据(Spatial data)映射到 “共享空间(Shared space)”,通过聚类(Cluster)对齐,生成 “单细胞分辨率的空间数据(Aligned single-cell resolution spatial data)”,划分 A、B、C 等区域。
核心逻辑
这是空间转录组与单细胞数据整合的典型方法,解决 **“单细胞类型的空间定位”** 问题 —— 先通过去卷积解析混合信号中的细胞类型比例,再通过映射(Mapping)将细胞类型与空间位置关联,最终构建 “细胞类型 - 空间分布” 图谱,常用于肿瘤微环境、发育生物学等研究。
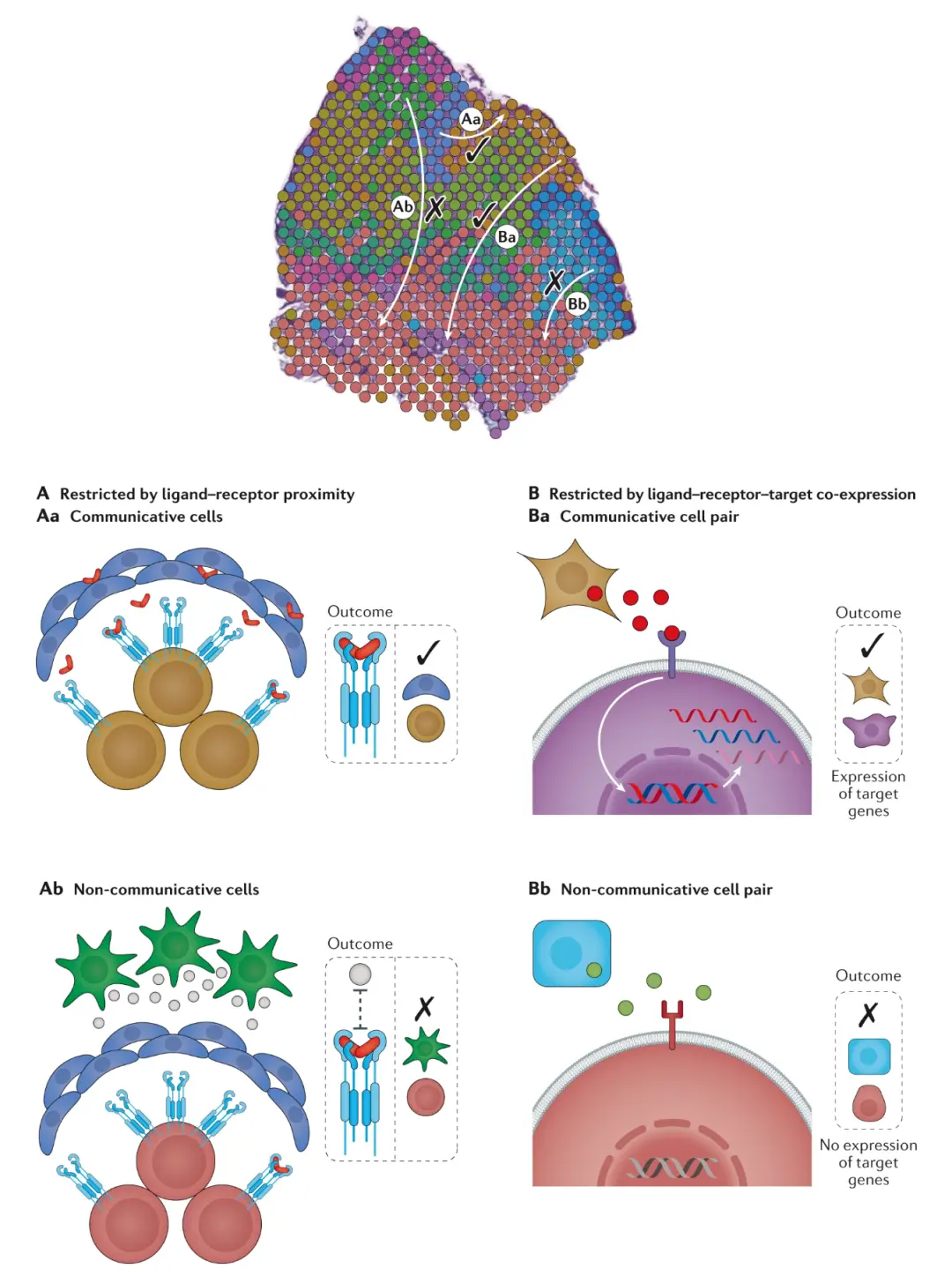
空间转录组学中细胞间通讯机制的示意图
核心模块
- 上方组织图谱:标注不同细胞通讯状态(Aa/Ba 为可通讯,Ab/Bb 为不可通讯 ),体现细胞通讯的空间特异性。
- 左侧(A):受 “配体 - 受体 proximity(空间邻近性)” 限制的细胞通讯:
- Aa:细胞空间邻近 + 配体 - 受体结合→可通讯(Outcome:√ );
- Ab:细胞空间分散 / 配体 - 受体不结合→不可通讯(Outcome:× )。
- 右侧(B):受 “配体 - 受体 - 靶基因共表达” 限制的细胞通讯:
- Ba:配体结合受体后,靶基因激活表达→可通讯(Outcome:√ );
- Bb:配体 - 受体未触发靶基因表达→不可通讯(Outcome:× )。
价值
这是空间细胞通讯分析的典型模型,解释 **“为什么有些细胞在空间上邻近却无法通讯”** —— 除了物理距离,还需满足 “配体 - 受体结合” 和 “靶基因激活” 等条件,为解析肿瘤微环境、发育信号通路提供理论框架。
详细文章链接:
热点综述 | 单细胞+空间转录组的整合分析方法总结_空间转录组细胞通讯方法-CSDN博客
单细胞转录组+空间转录组联合应用,1+1>2-CSDN博客
)








)
)








)