【图书推荐】《R语言医学数据分析实践》-CSDN博客
《R语言医学数据分析实践 李丹 宋立桓 蔡伟祺 清华大学出版社9787302673484》【摘要 书评 试读】- 京东图书 (jd.com)
预测ICU患者死亡率对比较药物的疗效、比较护理的有效性、比较手术的有效性有重要意义,利用机器学习来构建预测模型,辅助临床预测有着重要的意义。
6.10.1 数据集背景
本数据集来自医学公开数据库MIMIC III ICU中的数据,有4000患者。纳入的指标除了RecordID住院号,年龄age,性别Gender,身高baseWeight ,体重ICU类别baseICUType,SAPS评分,SOFA评分,住院时间 Length_of_stay , Surivial生存天数,院内死亡In.hospital_death。
其他指标处理,根据临床的相关性选择纳入的指标,这些纳入的指标在机器学习中的术语叫特征feature。比如根据临床经验,格拉斯哥昏迷评分“GCS”的最低值min及中位数mean可能预测死亡。其它指标也按照“GCS”同样处理,最后纳入的指标有94项,如图6-19所示。当然某项临床指标是否纳入,如何纳入,没有明确答案,靠临床经验和业务知识。

图6-19
6.10.2 数据预处理
原始数据中的某些指标在某些患者中没有观测值,发现很多缺失值,数据表中以“NA”表示。读入数据,查看每一列数据概况,R代码如下所示:
#数据文件icumortality.csv的路径根据实际情况修改sur<-read.csv("c:/icu_demo/icumortality.csv", header = T, sep = ",")summary(sur)查看每一列数据概况,summary函数会输出每一列数据平均值、最大值、最小值以及缺失情况,如图6-20所示,由于结果输出太长,这里截图并不完整。

图6-20
比如患者的性别Gender 值是0或1,有些患者值为-1,明显是推断错误,可以改为1。有些患者的height身高值小于100,weight体重小于10,也是推断不可能,可以赋值NA。计算患者的体重指数BMI,计算完再删除身高和体重两列。患者minTemp,meanTemp体温小于33°,PH值minPH 小于6或maxPH大于8,也是不可能,可以都赋值“NA”。
如图6-21所示,medianTroponinI缺失值达3795个(90%以上),可以删除,暂时不用的RecordID,SAPS.I、SOFA、length_of_stay、survival可以删除。

图6-21
MechVent指标是机械通气,0表示无,1表示有,如图6-22所示,发现部分患者MechVent值为“NA”,推断没有机械通气,可以赋值为0。

图6-22
血压有有创血压和无创血压两种,可以先找出有创血压DiasABP,SysABP 、MAP的缺失值,用此患者的的无创血压(带NI的变量)替换,然后删除无创血压的数据。
以上是利用经验判断缺失值是什么,此外可以使用mice程序包进行插补。mice即是基于多重填补法构造的,基本思想是对于一个具有缺失值的变量,用其他变量的数据对这个变量进行拟合,再用拟合的预测值对这个变量的缺失值进行填补。
数据清洗R代码如下:
#数据清洗
sur$baseGender[sur$baseGender<0] <- 1
sur$baseHeight[which(sur$baseHeight < 100)] <- NA
sur$baseHeight[which(sur$baseHeight > 210)] <- NA
sur$baseWeight[which(sur$baseWeight < 10)] <- NA
sur$BMI <- (sur$baseWeight/sur$baseHeight)/sur$baseHeight * 10000
sur[,"baseWeight"] <- NULL
sur[,"baseHeight"] <- NULL
sur[,"medianTroponinI"] <- NULLsub=which(is.na(sur$meanDiasABP))
sur$meanDiasABP[sub] <- sur$meanNIDiasABP[sub]
sur$minDiasABP[sub] <- sur$minNIDiasABP[sub]
sur$maxDiasABP[sub] <- sur$maxNIDiasABP[sub]
sur[,"meanNIDiasABP"] <- NULL
sur[,"minNIDiasABP"] <- NULL
sur[,"maxNIDiasABP"] <- NULL
sub=which(is.na(sur$meanMAP))
sur$meanMAP[sub] <- sur$meanNIMAP[sub]
sur$minMAP[sub] <- sur$minNIMAP[sub]
sur$maxMAP[sub] <- sur$maxNIMAP[sub]
sur[,"meanNIMAP"] <- NULL
sur[,"minNIMAP"] <- NULL
sur[,"maxNIMAP"] <- NULL
sub=which(is.na(sur$meanSysABP))
sur$meanSysABP[sub] <- sur$meanNISysABP[sub]
sur$minSysABP[sub] <- sur$minNISysABP[sub]
sur$maxSysABP[sub] <- sur$maxNISysABP[sub]
sur[,"meanNISysABP"] <- NULL
sur[,"minNISysABP"] <- NULL
sur[,"maxNISysABP"] <- NULLsur$minTemp[sur$minTemp < 33] <- NA
sur$meanTemp[sur$meanTemp < 33] <- NAsur$minpH[sur$minpH>8] <- NA
sur$minpH[sur$minpH<6] <- NA
sur$meanpH[sur$meanpH>8] <- NA
sur$maxpH[sur$maxpH>8] <- NAsur$meanMechVent[is.na(sur$meanMechVent)] <- 0sur[,"RecordID"] <- NULL
sur[,"SAPS.I"] <- NULL
sur[,"SOFA"] <- NULL
sur[,"Length_of_stay"] <- NULL
sur[,"Survival"] <- NULL
对于缺失值插补,可以利用mice程序包进行插补:
library(mice)
sur_clean <- mice(sur, m=5, seed=123456)
completedData <- complete(sur_clean,1)
6.10.3 模型建立
首先我们先用大家熟悉的Logistic回归分析模型,载入glmnet包,这个R包包括广义线性回归方程所有功能,R代码如图6-23所示:

图6-23
R代码运行,结果如图6-24所示。

图6-24
因为我们最后要比较各个机器学习模型的表现,需要新建一个表格用来存储模型的AUC,新建一个表格final,新表格只有一列label,是真实的死亡情况,新表格增加一列lasso,存储lasso模型的预测值,R代码如下所示:
label<-completedData$In.hospital_death
final <- as.data.frame(label)
final$Lasso <- completedData$test_pred
我们接着看支持向量机SVM ,给定一组训练样本集,样本数据集是二维的,分散在平面上,需要找到一条直线将数据集分割开。可以分开的直线有很多,我们要找到其中泛化能力最好,鲁棒性最强的直线。这是在平面上的点,如果是在三维空间中,则需要找到一个平面;如果是超过三维以上的维数,则需要找到一个超平面。
SVM的三个重要参数是kernel参数、C参数cost和gamma参数来调节模型的表现:
(1)C参数:C参数(Cost)它是控制着模型的惩罚力度,即决定了模型在训练时对误差和复杂度之间的折衷关系。通常情况下,C值越大,模型的复杂度越高,预测准确度也相应提高。但是,如果C值过大,可能会导致模型出现过拟合的情况,因此需要在实际应用中进行试探和调整。
(2)gamma参数:gamma参数决定了样本点对于模型的影响程度,即越接近样本点的数据在模型中的权重也越大。通常情况下,gamma值越小,对于远离样本点的数据的影响也就越小,而对于靠近样本点的数据的影响则更加强烈。但是,如果gamma值过小,可能会导致模型出现欠拟合的情况,因此需要进行合理的选择。
(3)kernel参数:Kernel指的是支持向量机的类型,它可能是线性还是非线性,首先选择线性的kernel作为基准。核函数是支持向量机回归算法的核心,不同的核函数适用于不同的数据集。常见的核函数有线性核函数、多项式核函数和径向基核函数等。在选择核函数时需要考虑数据集的特点和所需的预测准确度。
C参数即Cost是违反约束时的成本函数,gamma是除线性SVM外其余所有SVM都使用的一个参数,它的选择与直线或平面的非线性程度相关,gamma选择越大,其非线性程度越大。
使用支持向量机模型R代码如图6-25所示。

代码运行,结果如图6-26所示。

图6-26
在final数据表中存储SVM模型的AUC
final$SVM <- completedData$test_pred随机森林Radom Forest算法介绍:简单的说,随机森林就是用随机的方式建立一个森林,森林里面有很多的决策树,并且每棵树之间是没有关联的。得到一个森林后,当有一个新的样本输入,森林中的每一棵决策树会分别进行一下判断,进行类别归类(针对分类算法),最后比较一下被判定哪一类最多,就预测该样本为哪一类。在随机森林方法中,创建了大量的决策树。每个观察结果都被送入每个决策树。 每个观察结果最常用作最终输出。对所有决策树进行新的观察,并对每个分类模型进行多数投票。
随机森林Radom Forest的模型R代码,如图6-27所示:

运行结果,如图6-27所示
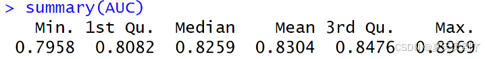
图6-27
同样,在final数据表中存储randomforest模型的AUC:
final$randomforest <- completedData$test_pred6.10.4 模型评估
利用基于plotROC包和ggplot2软件包可以将模型得到的AUC都展示在同一张ROC曲线图中,这样比较直观比较。如果提示出现“Error in library(plotROC) : 不存在叫‘plotROC’这个名字的程辑包”,可以通过 “install.packages("plotROC", dependencies = TRUE)”命令安装plotROC包。
R代码如下所示:
library(plotROC)
library(ggplot2)
longtest <- melt_roc(final, "label", c("Lasso", "randomforest"))
cbPalette <- c("#FF0000", "#FFCC00")
cbPalette <- c("#FF0000", "#FFCC00")
ggplot(longtest, aes(d = D, m = M, color = name)) +scale_colour_manual(values=cbPalette) + geom_roc(size = 1.75, n.cuts = 0, labels = FALSE) + style_roc() + theme(axis.text.x= element_text(size = 22), axis.title.x= element_text(size = 22), axis.text.y= element_text(size = 22), axis.title.y= element_text(size = 22), legend.text = element_text(size = 22))
运行结果,如图6-28所示。可以看出随机森林的AUC最大。

图6-28


(TCP的连接管理、可靠性、面临复杂网络的处理))


)


)


-----深度优先搜索(DFS)实现)








